構造生物学研究室
概要
X線回折・散乱法や単粒子低温電子顕微鏡法などを用いて、細胞運命の本体であるDNAメチル化の形成と継承、ストレス応答や脂質代謝を制御する膜上でのシグナル伝達に関与するタンパク質や核酸(DNA, RNA)及びそれらの集合体の構造を決定し、これらの分子の生体内での働きを解析するとともに、その異常な振る舞いよって引き起こされる疾患の仕組みを構造科学的に明らかにする。
スタッフ

<略歴>
横浜市立大学大学院総合理学研究科博士後期課程修了(2006年)理学博士。2005年日本学術振興会特別研究員、2010年から京都大学工学研究科助教。2013横浜市立大学大学院生命医科学研究科准教授。 2021年4月から同大学院教授。
<メッセージ>
研究の1番の醍醐味は、誰も今まで見たこともない現象を誰よりも早く目の当たりにできることです。構造生物学は、生体分子の形を明らかにして生命現象の謎を紐解く研究分野です。研究を楽しんで心躍るような成果をあげてみませんか?我々の研究室では多くの学生が活躍し研究に励んでいます。是非一緒に世界を あっ! と言わせるような研究をしましょう。
横浜市立大学大学院総合理学研究科博士後期課程修了(2006年)理学博士。2005年日本学術振興会特別研究員、2010年から京都大学工学研究科助教。2013横浜市立大学大学院生命医科学研究科准教授。 2021年4月から同大学院教授。
<メッセージ>
研究の1番の醍醐味は、誰も今まで見たこともない現象を誰よりも早く目の当たりにできることです。構造生物学は、生体分子の形を明らかにして生命現象の謎を紐解く研究分野です。研究を楽しんで心躍るような成果をあげてみませんか?我々の研究室では多くの学生が活躍し研究に励んでいます。是非一緒に世界を あっ! と言わせるような研究をしましょう。
准教授 禾 晃和(のぎ てるかず) ResearchMap → 禾グループWEBサイト→

<略歴>
京都大学大学院理学研究科化学専攻博士課程修了(2001年)理学博士。2001年高エネ研物構研・博士研究員。2002年マックスプランク生物物理研・博士研究員。2004年阪大蛋白研・助手(2007年助教)。2011年4月から横浜市立大学准教授。
<メッセージ>
構造生物学は、生物学、化学、物理学など、色々なバックグラウンドを持った人間がそれぞれの得意分野を生かして研究に取り組むことができる領域です。我々のグループでは、タンパク質分子が外界からの情報を受け取り、細胞の中へと受け渡している姿を結晶や溶液の中で再現し、原子・分子のレベルで可視化することを目指して研究を進めています。
京都大学大学院理学研究科化学専攻博士課程修了(2001年)理学博士。2001年高エネ研物構研・博士研究員。2002年マックスプランク生物物理研・博士研究員。2004年阪大蛋白研・助手(2007年助教)。2011年4月から横浜市立大学准教授。
<メッセージ>
構造生物学は、生物学、化学、物理学など、色々なバックグラウンドを持った人間がそれぞれの得意分野を生かして研究に取り組むことができる領域です。我々のグループでは、タンパク質分子が外界からの情報を受け取り、細胞の中へと受け渡している姿を結晶や溶液の中で再現し、原子・分子のレベルで可視化することを目指して研究を進めています。
研究内容
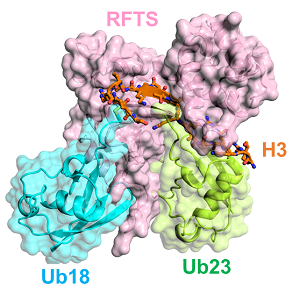 DNMT1 RFTSドメインとK18とK23がユビキチン化されたヒストンH3の複合体構造 (Isiyama et al., 2017)
DNMT1 RFTSドメインとK18とK23がユビキチン化されたヒストンH3の複合体構造 (Isiyama et al., 2017)
(有田グループ) DNAメチル化は細胞の遺伝子発現パターンを定義し、細胞運命を決める重要なエピジェネティックな情報です。ゲノム配列と同様に、DNAメチル化は複製・細胞分裂を経て次世代の細胞に正確に継承されます。正常な細胞の増殖にはDNA維持メチル化の機構は必須であり、その破たんはがんなどの様々な病気を引き起こします。DNA維持メチル化に関与する生体分子に着目し、その立体構造をX線結晶構造解析、X線溶液散乱、高速AMF,クライオ電子顕微鏡を用いて決定することで、DNA維持メチル化の基本原理の解明と維持メチル化因子を標的とした阻害剤の開発を目指します。
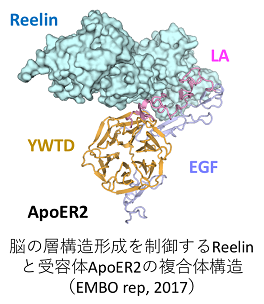
(禾グループ)細胞膜上において1回膜貫通型タンパク質が行うシグナル伝達を取り上げ、細胞外からのシグナルの受容と膜を隔てた細胞内へのシグナル伝達の仕組みを分子・原子のレベルで理解することを目的として構造研究を進める。脳神経の発生に関わるシグナル伝達や細菌における膜内配列切断を介したシグナル伝達を取り上げ、1回膜貫通型の受容体やシグナル分子が、細胞膜という二次元の反応場において、弱い相互作用ながらも精巧な分子認識を行い、ダイナミックな構造変化を起こしてシグナルを変換する様子を、X線結晶解析や溶液散乱、電子顕微鏡イメージング等の構造解析手法を複合的に用いて捉えていく。
主要文献(Selected Publications)
‣Tamura-Sakaguchi R, Aruga R, Hirose M, Ekimoto T, Miyake T, Hizukuri Y, Oi R, Kaneko MK, Kato Y, Ikeguchi M, AkiyamaY, Iwasaki K, Nogi T.* Moving toward generalizable NZ-1 labeling for 3D structure determination with optimized epitope tag insertion. Acta Cryst D (2021)
‣Kori S, Jimenji T, Ekimoto T, Sato M, Kusano F, Oda T, Unoki M, Ikeguchi M, Arita K. Serine 298 phosphorylation in linker 2 of UHRF1 regulates ligand-binding property of its tandem Tudor domain. J. Mol. Biol. 432 4061-4075. (2020)
‣Nishiyama A*, Mulholland C, Bultmann S, Kori S, Endo A, Saeki Y, Qin W, Trummer C, Chiba Y, Yokoyama H, Kumamoto S, Kawakami T, Hojo H, Nagae G, Aburatani H, Tanaka K, Arita K*, Leonhardt H*, and Nakanishi M*. Two distinct modes of DNMT1 recruitment ensure stable maintenance DNA methylation. Nature Communications. 11, 1222. (2020)
‣Tamura R, Oi R, Akashi S, Kaneko MK, Kato Y, Nogi T. Application of the NZ-1 Fab as a crystallization chaperone for PA tag-inserted target proteins. Prot Sci 28, 823-836 (2019)
‣Jimenji T, Matsumura R, Kori S, Arita K. Structure of PCNA in complex with DNMT1 PIP box reveals the basis for the molecular mechanism of the interaction. Biochem Biophys Res Commun. 516 578-583. (2019)
‣Kori S, Ferry L, Matano S, Jimenji T, Kodera N, Tsusaka T, Matsumura R, Oda T, Sato M, Dohmae N, Ando T, Shinkai Y, Defossez PA*, Arita K*. Structure of the UHRF1 Tandem Tudor Domain Bound to a Methylated Non-histone Protein, LIG1, Reveals Rules for Binding and Regulation. Structure 27, 485-496 (2019)
‣Nogi T*. How multi-scale structural biology elucidated context-dependent variability in ectodomain conformation along with the ligand capture and release cycle for LDLR family members, Bioplys Rev 10, 481-492 (2018)
‣Ishiyama S, Nishiyama A, Saeki Y, Moritsugu K, Morimoto D, Yamaguchi L, Arai N, Matsumura R, Kawakami T, Mishima Y, Hojo H, Shimamura S, Ishikawa F, Tajima S, Tanaka K, Ariyoshi M, Shirakawa M, Ikeguchi M, Kidera A, Suetake I, Arita K*, Nakanishi M*. Structure of the Dnmt1 reader module complexed with a unique two-mono-ubiquitin mark on histone H3 reveals the basis for DNA methylation maintenance. Mol Cell 68, 350-360 (2017)
‣Hirai H, Yasui N, Yamashita K, Tabata S, Yamamoto M, Takagi J, and Nogi T*. Structural basis for ligand capture and release by the endocytic receptor ApoER2, EMBO rep 18, 982-999 (2017)
‣Kori S, Jimenji T, Ekimoto T, Sato M, Kusano F, Oda T, Unoki M, Ikeguchi M, Arita K. Serine 298 phosphorylation in linker 2 of UHRF1 regulates ligand-binding property of its tandem Tudor domain. J. Mol. Biol. 432 4061-4075. (2020)
‣Nishiyama A*, Mulholland C, Bultmann S, Kori S, Endo A, Saeki Y, Qin W, Trummer C, Chiba Y, Yokoyama H, Kumamoto S, Kawakami T, Hojo H, Nagae G, Aburatani H, Tanaka K, Arita K*, Leonhardt H*, and Nakanishi M*. Two distinct modes of DNMT1 recruitment ensure stable maintenance DNA methylation. Nature Communications. 11, 1222. (2020)
‣Tamura R, Oi R, Akashi S, Kaneko MK, Kato Y, Nogi T. Application of the NZ-1 Fab as a crystallization chaperone for PA tag-inserted target proteins. Prot Sci 28, 823-836 (2019)
‣Jimenji T, Matsumura R, Kori S, Arita K. Structure of PCNA in complex with DNMT1 PIP box reveals the basis for the molecular mechanism of the interaction. Biochem Biophys Res Commun. 516 578-583. (2019)
‣Kori S, Ferry L, Matano S, Jimenji T, Kodera N, Tsusaka T, Matsumura R, Oda T, Sato M, Dohmae N, Ando T, Shinkai Y, Defossez PA*, Arita K*. Structure of the UHRF1 Tandem Tudor Domain Bound to a Methylated Non-histone Protein, LIG1, Reveals Rules for Binding and Regulation. Structure 27, 485-496 (2019)
‣Nogi T*. How multi-scale structural biology elucidated context-dependent variability in ectodomain conformation along with the ligand capture and release cycle for LDLR family members, Bioplys Rev 10, 481-492 (2018)
‣Ishiyama S, Nishiyama A, Saeki Y, Moritsugu K, Morimoto D, Yamaguchi L, Arai N, Matsumura R, Kawakami T, Mishima Y, Hojo H, Shimamura S, Ishikawa F, Tajima S, Tanaka K, Ariyoshi M, Shirakawa M, Ikeguchi M, Kidera A, Suetake I, Arita K*, Nakanishi M*. Structure of the Dnmt1 reader module complexed with a unique two-mono-ubiquitin mark on histone H3 reveals the basis for DNA methylation maintenance. Mol Cell 68, 350-360 (2017)
‣Hirai H, Yasui N, Yamashita K, Tabata S, Yamamoto M, Takagi J, and Nogi T*. Structural basis for ligand capture and release by the endocytic receptor ApoER2, EMBO rep 18, 982-999 (2017)