|
峔憿惗暔妛幚廗2003偵偍偗傞Xfit偺巊梡朄 嫶杮丂攷 XtalView偼D. M. McRee巵偵傛偭偰奐敪偝傟偨僞儞僷僋幙寢徎峔憿夝愅傪偍偙側偆偨傔偺僾儘僌儔儉僷僢働乕僕偺堦偮偱偁傞丅寢徎峔憿夝愅偵昁梫側僜僼僩僂僄傾偼傎偲傫偳娷傑傟偰偄傞丅 崱夞偺幚廗偱偼丄XtalView偵娷傑傟傞僌儔僼傿僢僋僜僼僩Xfit傪巊梡偟丄Lysozyme傪懳徾偵倃慄寢徎峔憿夝愅偵偍偗傞僞儞僷僋幙暘巕儌僨儖偺峔抸傪懱尡偡傞丅 棳傟 儕僝僠乕儉暘巕偼129傾儈僲巁偐傜峔惉偝傟偰偄傞丅揹巕枾搙傪尦偵峔抸偟偨晹暘峔憿乮億儕傾儔僯儞偺傊儕僢僋僗乯傪弶婜儌僨儖偲偡傞丅 ------------------------------------------------------------- 1.弨旛 儂乕儉僨傿儗僋僩儕偵嶌嬈僨傿儗僋僩儕傪偮偔傞丅 % cd 嶌嬈僨傿儗僋僩儕偵幚廗偵昁梫側嵗昗(PDB)偲揹巕枾搙(MAP)僼傽僀儖傪僐僺乕偡傞丅 % cd xtalview --------------------亙栠傞亜---亙師傊亜----------------------- 2.揹巕枾搙偲晹暘峔憿乮兛傊儕僢僋僗偺億儕傾儔僯儞儌僨儖乯偺昞帵
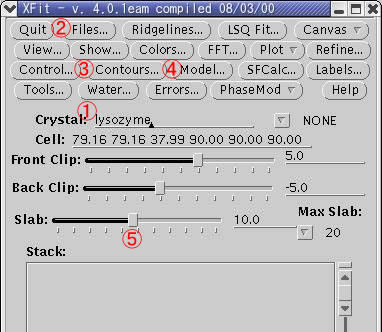
% xfit 傪幚峴偡傞偲丄Xfit偺儊僯儏乕僂僀儞僪僂偲恀偭崟側僂僀儞僪僂偑昞帵偝傟傞丅 Xfit偺儊僯儏乕僂僀儞僪僂 嘆lysozyme偲擖椡偟Enter僉乕傪墴偡丅乮奿巕掕悢偲嬻娫孮偑撉傒崬傑傟傞丂乯
丂
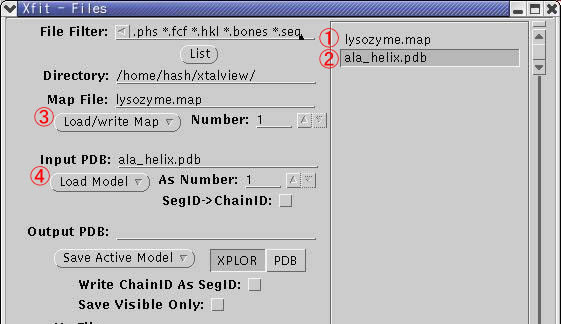
嘆嘇 lysozyme.map偲ala_helix.pdb傪塃僋儕僢僋偡傞偲僼傽僀儖偑慖戰偝傟傞丅
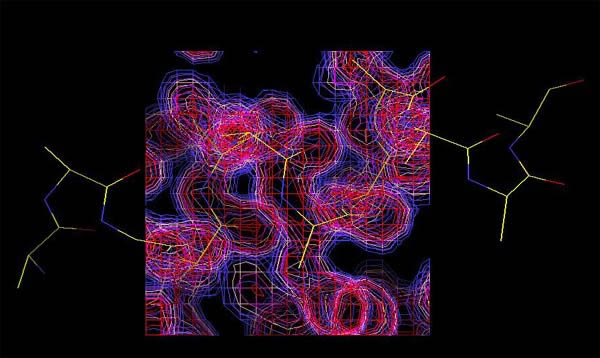
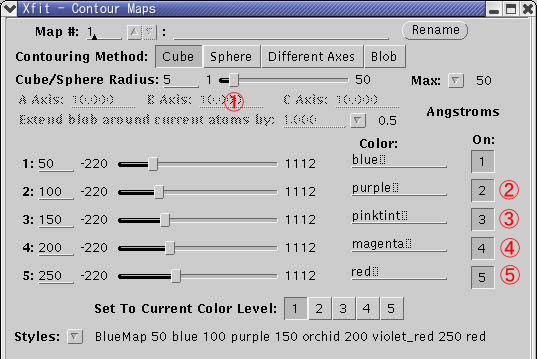
昞帵偝傟偰偄傞暘巕偼丄扽慺尨巕偑墿怓丄拏慺尨巕偑惵怓丄巁慺尨巕偑愒怓偱昞尰偝傟偰偄傞丅乮悈慺尨巕偼敀丄棸墿尨巕偼椢乯 Xfit偺儊僯儏乕僂僀儞僪僂偱Contours...傪嵍僋儕僢僋偡傞偲揹巕枾搙偺昞帵傪曄峏偡傞Contour僂僀儞僪僂偑尰傟傞丅 丂 嘆偺儗儀儖乮仩乯傪嵍塃偵摦偐偡偙偲偱丄昞帵偝傟傞揹巕枾搙偺斖埻傪曄偊傞偙偲偑偱偒傞丅
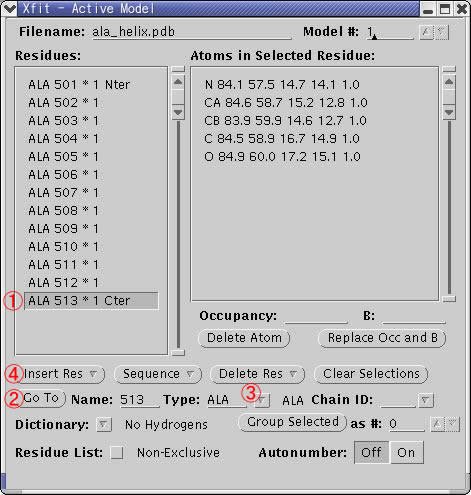
Xfit偱暘巕傒傞曽朄乮儅僂僗偺憖嶌朄乯 嵍僋儕僢僋偟側偑傜儅僂僗傪摦偐偡偲暘巕偑夞揮偡傞丅 Xfit偺儊僯儏乕僂僀儞僪僂偱Model....傪嵍僋儕僢僋偡傞偲尰嵼撉傒崬傑傟偰偄傞峔憿偺忣曬傪傒傞偙偲偑偱偒傞丅 嵍懁偺Residues:偺棑偵偼丄尰嵼昞帵偝傟偰偄傞儌僨儖偺攝楍偲巆婎斣崋偑昞帵偝傟偰偄傞丅
丂 --------------------亙栠傞亜---亙師傊亜----------------------- 儕僝僠乕儉偺傾儈僲巁攝楍偐傜偺擇師峔憿梊應 > Lysozyme - chicken egg
white - 129 aa,丂Molecular Weight 14 kDa 擇師峔憿梊應偼埲壓偺僒僀僩傪棙梡偟偨 http://bioinf.cs.ucl.ac.uk/psipred/psiform.html
尰嵼昞帵偝傟偰偄傞揹巕枾搙偲億儕傾儔僯儞儌僨儖傪堦偮偢偮尒偰丄懁嵔偺揹巕枾搙偺摿挜傪攃埇偡傞丅 懁嵔偺抲姺曽朄 Xfit偺儊僯儏乕僂僀儞僪僂偱Model....傪嵍僋儕僢僋 嘆抲姺偟偨偄巆婎傪慖戰 偙偺傛偆偵偟偰501偐傜513傑偱偺傾儔僯儞傪幚嵺偺傾儈僲巁偵抲偒姺偊偰偄偔丅
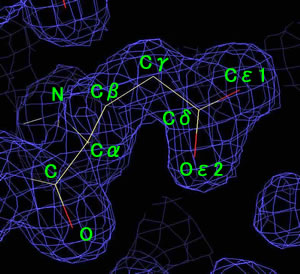
僸儞僩丗 僶儕儞偺揹巕枾搙偵丄岆偭偰僸僗僠僕儞偵抲姺偟偨椺 僸僗僠僕儞偺懁嵔偑丄揹巕枾搙偐傜偼傒弌偰偍傝丄僼傿僢僩偝偣傞偙偲偑偱偒側偄丅
僌儖僞儈儞巁偺懁嵔傪揹巕枾搙偵僼傿僢僩偝偣偨椺 惓偟偄傾儈僲巁偵抲姺偡傞偲丄壓恾偺傛偆偵丄傾儈僲巁偺懁嵔偑揹巕枾搙偵偒偭偪傝偲僼傿僢僩偡傞丅
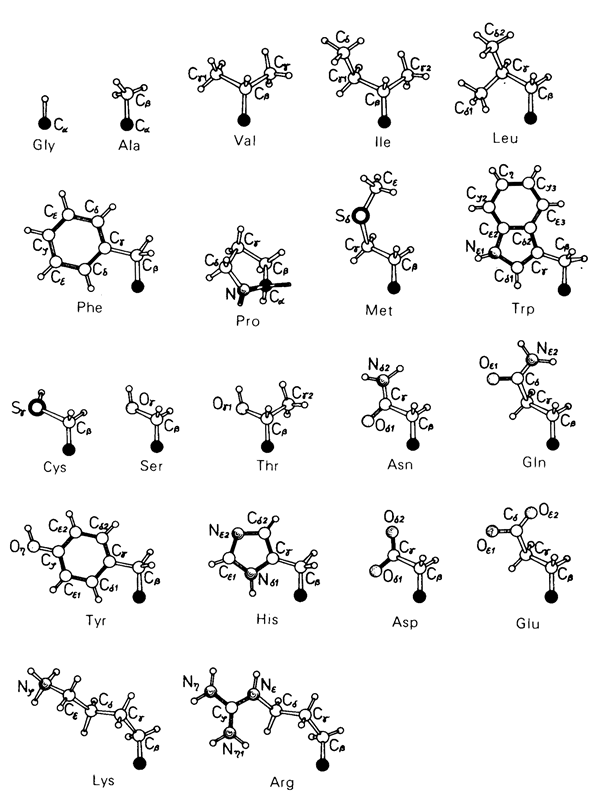
丂 俀侽庬椶偺傾儈僲巁偺峔憿 丂
|