|
!!2006年の実習からCOOTを使ったモデリングに変更しています!!
COOTのマニュアルはこちら
構造生物学実習 2005
Last update
2008-06-12 9:30
早く終わった人は、、、
-----------------------------------------------------------------------------
 はじめに はじめに
X線結晶構造解析では、電子密度とよばれる電子の分布図をもとにタンパク質分子の構造を決定する。電子密度は電子の存在位置をしめしたものであり、そこには原子が存在すると考えれば、電子密度に沿って各原子を並べていくことで、巨大な蛋白質分子の構造を決定することができる。しかし、タンパク質はアミノ酸がペプチド結合でつながったものであるから、実際には原子ではなくアミノ酸を並べていく。通常は初期電子密度から対象とする蛋白質のポリアラニンモデルを作成する。
 電子密度と分解能 電子密度と分解能
分解能(resolution, 解像度)によって、電子密度の見え方は全く異なる。
3.0Å分解能程度の電子密度を得ることができれば、分子構造を解析することができる。
今回の実習では、プログラムXtalViewに含まれるグラフィックソフトXfitを使用し、リゾチーム分子を構築する。
プログラムXtalViewはD. M. McRee氏によって開発されたタンパク質結晶構造解析を行うためのプログラムパッケージの一つである。XtalViewの入手に関しては以下のサイトを参照。
http://www.scripps.edu/pub/dem-web/toc.html
リゾチーム分子は129アミノ酸から構成されている。電子密度を元に構築した部分構造(ポリアラニンのへリックス)を初期モデルとする。
電子密度を見ながら、アラニンから実際のアミノ酸に置き換えていき、このヘリックスが、リゾチーム分子のどこの部分に相当するかを考える。
 準備 準備
Linuxでログインし、ホームディレクトリに作業ディレクトリをつくる。
% cd
% mkdir xtalview
作業ディレクトリに実習に必要な座標(PDB)と電子密度(MAP)ファイルをコピーする。
% cd xtalview
% cp /home/share/xray/2005/ala_helix.pdb .
% cp /home/share/xray/2005/lysozyme.map .
 電子密度と部分構造(αへリックスのポリアラニンモデル)の表示 電子密度と部分構造(αへリックスのポリアラニンモデル)の表示
作業ディレクトリで
% xfit
を実行すると、XfitのMenu Window(図1)と真っ黒なウインドウが表示される。
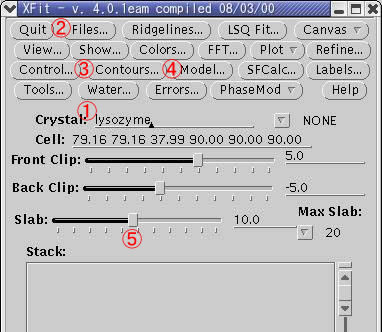
図1
Menu Window
①lysozymeと入力しEnterキーを押す。(格子定数と空間群が読み込まれる )
②Files...を左クリックすると、電子密度(マップ)や座標(PDB)を読み込ませるFiles Window(図2)が現れる。
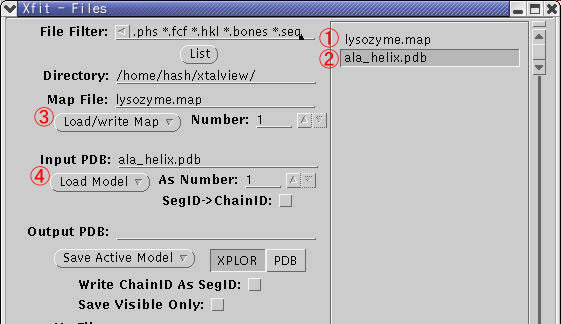
図2
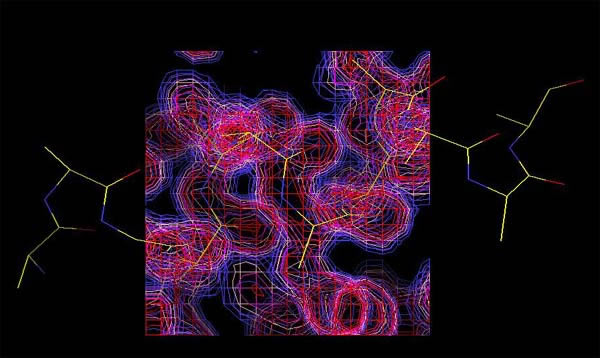
図3 ポリアラニンモデルと電子密度
表示されている分子は、炭素原子が黄色、窒素原子が青色、酸素原子が赤色で表現されている。(水素原子は白、硫黄原子は緑)
XfitのMenu Window(図1)で③Contours...を左クリックすると電子密度の表示を変更するContour Windowが現れる(図4)。
Contour Window
① のレベル(□)を左右に動かすことで、表示される電子密度の範囲を変えることができる。
6くらい表示すれば十分である。(あまり表示範囲を大きくしてしまうと、動きが遅くなる)
表示されている電子密度は、その強度レベルに応じて5段階に色分けされている。通常レベル1のみを表示するので、ほかの2 - 4のレベルは表示しない。②~⑤を左クリックして、レベル2から5の電子密度が表示されないようにする。
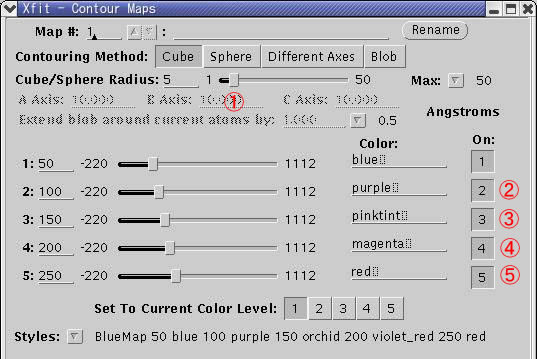
図4
 Xfitで分子を見る方法(マウスの操作法) Xfitで分子を見る方法(マウスの操作法)
左クリックしながらマウスを動かすと分子が回転する。
中クリックしながらマウスを動かすと分子が並進する。
左中を同時クリックしながらマウスを動かすと分子が拡大・縮小する。
XfitのメニューウインドウでModel....を左クリックするとActive Model Windowが開き、現在読み込まれている構造の情報をみることができる(図5)。
左側のResidues:の欄には、現在表示されているモデルの配列と残基番号が表示されている。
①例えばALA513を選択して(クリックして)、②Go Toを左クリックすればALA513のCαが画面の中心にくる。
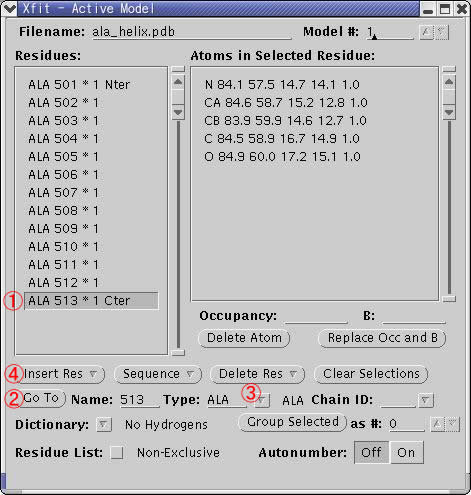
図5
 アミノ酸の置換 アミノ酸の置換
リゾチームのアミノ酸配列
> Lysozyme - chicken egg white - 129 aa, Molecular Weight 14 kDa
KVFGRCELAA AMKRHGLDNY RGYSLGNWVC
AAKFESNFNT QATNRNTDGS
TDYGILQINS RWWCNDGRTP GSRNLCNIPC SALLSSDITA SVNCAKKIVS
DGNGMNAWVA WRNRCKGTDV QAWIRGCRL
現在表示されている電子密度とポリアラニンモデルを一つずつ見て、側鎖の電子密度の特徴を把握する。アミノ酸配列を参考に、正しいと思われるアミノ酸に置き換える。
側鎖の置換方法
Menu Window(図1)でModel....を左クリックしActive Model Windowを開く(図5)。
①置換したい残基を選択
③▽を右クリックするとアミノ酸のリストが現れるので、置き換えるアミノ酸を選ぶ
④Insert Resを右クリックし、Replace and Fitを選択すると、アミノ酸が置換され、かつ自動で電子密度にフィットする。
置き換えた後に、側鎖が電子密度にフィットできない場合は、間違ったアミノ酸に置き換えた可能性が高いので、他のアミノ酸を検討する。
このようにして501から513までのアラニンを実際のアミノ酸に置き換えていく。
ヒント:
・比較的大きな側鎖(芳香族アミノ酸など)に注目する。
・20種類のアミノ酸の構造を参考にしながら、側鎖を推測する。
・とにかく、特徴的な(大きな)側鎖の電子密度を持つものを、他のアミノ酸に置換してみる。明らかに間違っているかどうかは、結果を見れば一目瞭然である。
・水素原子は無視する。
・電子密度からだけでは、グルタミン酸とグルタミン、アスパラギン酸とアスパラギンを区別することは難しい。アミノ酸配列から判定するしかない。
・二次構造予測を参考にする
例えば http://bioinf.cs.ucl.ac.uk/psipred/psiform.html
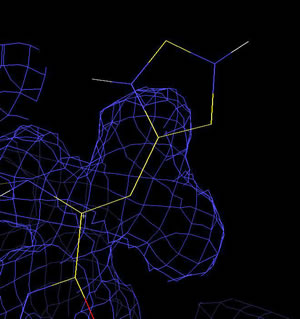
バリンの電子密度に、誤ってヒスチジンに置換した例
ヒスチジンの側鎖が、電子密度からはみ出ており、フィットさせることができない。
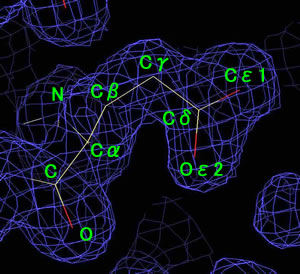
グルタミン酸の側鎖を電子密度にフィットさせた例
正しいアミノ酸に置換すると、下図のように、アミノ酸の側鎖が電子密度にきっちりとフィットする。
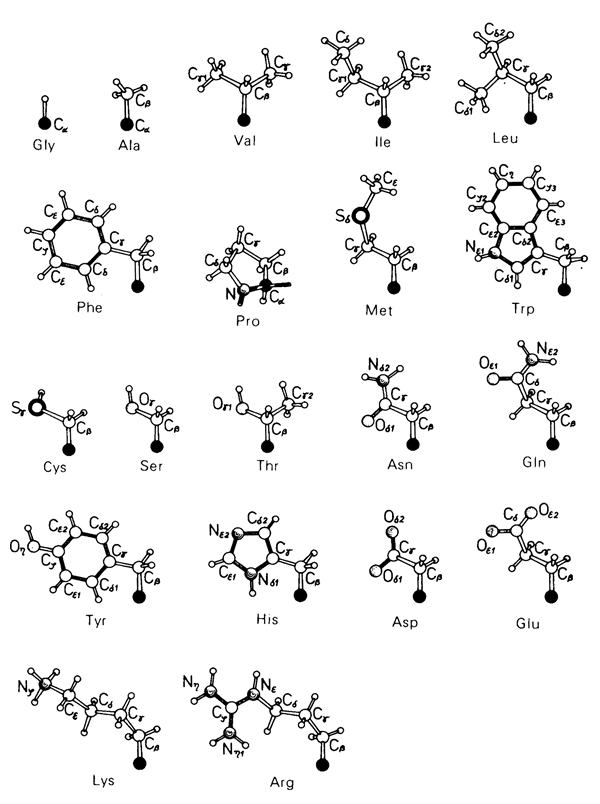
 20種類のアミノ酸の構造 20種類のアミノ酸の構造
(タンパク質 構造・機能・進化 G.E. Shlulz, R. H. Shirmer著 大井龍夫 監訳 化学同人 より転載)
 早く終わった人は、、、、 早く終わった人は、、、、
これ以外に5つのフラグメントを用意してあるので、それに挑戦してみる。
% cp /home/share/xray/2005/frag1.pdb .
% cp /home/share/xray/2005/frag2.pdb .
% cp /home/share/xray/2005/frag3.pdb .
% cp /home/share/xray/2005/frag4.pdb .
% cp /home/share/xray/2005/frag5.pdb .

|

