有田(恭)研究室のM1舩生智子さんの研究成果がJ.Biochemに掲載!
2021.08.11
- TOPICS
- 研究
- 学生の活躍
活性型ユビキチン化SETDB1の調製に成功!
発表者:舩生智子 (博士前期1年), 金丸由桂 (2020年度卒), 小野田浩宜 (特任助教: https://researchmap.jp/hiroki_onoda ), 有田 恭平 (教授: https://researchmap.jp/read0146480 )
研究分野: 構造生物学
キーワード: ヒストン修飾、ユビキチン化、エピジェネティクス、レトロトランスポゾン
構造生物学研究室 有田グループ (http://www-mls.tsurumi.yokohama-cu.ac.jp/xtal-mls/members/kyouhei/index.html )
研究分野: 構造生物学
キーワード: ヒストン修飾、ユビキチン化、エピジェネティクス、レトロトランスポゾン
構造生物学研究室 有田グループ (http://www-mls.tsurumi.yokohama-cu.ac.jp/xtal-mls/members/kyouhei/index.html )
研究成果のポイント
- ユビキチン化酵素とSETDB1を大腸菌内で共発現させて、部位特異的なSETDB1のユビキチン化に成功した。
- ユビキチン化SETDB1がヒストンメチル化活性を持つことを確認し、本研究の大腸菌内ユビキチン化システムの有用性を示した。
- 大腸菌内ユビキチン化システムはユビキチン化によるSETDB1の酵素活性化機構の解明を目指す構造生物学的や生化学的な研究の重要なツールとなる。
発表内容
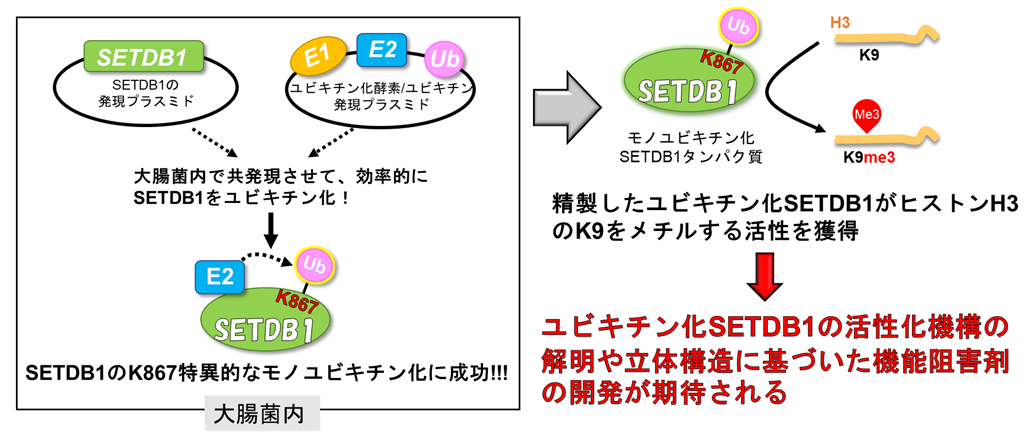 図 1 大腸菌内での酵素活性化型モノユビキチン化SETDB1の調製
図 1 大腸菌内での酵素活性化型モノユビキチン化SETDB1の調製
研究背景
哺乳類のゲノムの約40%は、動く遺伝子と呼ばれるトランスポゾン由来の配列で占められています。その中でも内因性レトロウイルス由来の配列は約10%を占めており、その異常な発現はゲノム不安定性を引き起こし、癌や自己免疫性疾患との関連が示唆されています。それに対抗する防御機構の一つとしてヒストンタンパク質の翻訳後修飾 (ヒストン修飾) が存在します。
真核生物の核内には、ゲノムDNAがコンパクトに折り畳まれた染色体が存在します。染色体は主にヒストンとDNAで構成され、これらが何重にも折り畳まったクロマチン構造をとります。クロマチン構造は主に2種類存在し、クロマチンが凝集し遺伝子発現がオフになる状態 (ヘテロクロマチン構造) と、クロマチンが緩み遺伝子発現がオンの状態 (ユークロマチン構造) があります。この遺伝子発現のオンとオフを担う因子の一つとしてヒストン修飾があります。ヒストン修飾は、ヒストンタンパク質の末端であるテールがメチル化・アセチル化・リン酸化・ユビキチン化などの翻訳後修飾されることであり、これらの修飾の種類やその組み合わせによって遺伝子発現が制御されます。
本研究対象であるSETDB1 (SET domain bifurcated histone lysine methyltransferase 1) はヒストンH3の9番目のリジン残基をトリメチル化する酵素であり、ヘテロクロマチン構造の形成を促進し、遺伝子発現または内因性レトロウイルスの転写を制御します。SETDB1のヒストンメチル化活性は、C末端にある保存されたSETドメインが担っており、SETドメイン内には約300アミノ酸からなる長いinsertionが存在しSETドメインを分断しています。興味深いことに、SETDB1のinsertion内の867番目のリジン (K867) がモノユビキチン化されることが、ヒストンメチル化活性に重要であることが明らかになっています。SETDB1は初期の発生や細胞分化、またその他の細胞機能において重要な役割を果たします。また、SETDB1の異常な発現は様々な疾患で見られ、特に癌において過剰発現しています。このように、SETDB1は様々な疾患に関連し生体内で重要な役割を担っているにも関わらず、その酵素活性化機構は未だに明らかになっていません。SETDB1は様々な疾患の治療標的であり、酵素活性化機構の解明は、SETDB1の活性を阻害する創薬の応用に繋がります。そこで本研究グループでは、SETDB1の酵素活性化機構を明らかにするための構造解析に向けた、大腸菌内での効率的な活性化型モノユビキチン化SETDB1の調製に取り組みました。
真核生物の核内には、ゲノムDNAがコンパクトに折り畳まれた染色体が存在します。染色体は主にヒストンとDNAで構成され、これらが何重にも折り畳まったクロマチン構造をとります。クロマチン構造は主に2種類存在し、クロマチンが凝集し遺伝子発現がオフになる状態 (ヘテロクロマチン構造) と、クロマチンが緩み遺伝子発現がオンの状態 (ユークロマチン構造) があります。この遺伝子発現のオンとオフを担う因子の一つとしてヒストン修飾があります。ヒストン修飾は、ヒストンタンパク質の末端であるテールがメチル化・アセチル化・リン酸化・ユビキチン化などの翻訳後修飾されることであり、これらの修飾の種類やその組み合わせによって遺伝子発現が制御されます。
本研究対象であるSETDB1 (SET domain bifurcated histone lysine methyltransferase 1) はヒストンH3の9番目のリジン残基をトリメチル化する酵素であり、ヘテロクロマチン構造の形成を促進し、遺伝子発現または内因性レトロウイルスの転写を制御します。SETDB1のヒストンメチル化活性は、C末端にある保存されたSETドメインが担っており、SETドメイン内には約300アミノ酸からなる長いinsertionが存在しSETドメインを分断しています。興味深いことに、SETDB1のinsertion内の867番目のリジン (K867) がモノユビキチン化されることが、ヒストンメチル化活性に重要であることが明らかになっています。SETDB1は初期の発生や細胞分化、またその他の細胞機能において重要な役割を果たします。また、SETDB1の異常な発現は様々な疾患で見られ、特に癌において過剰発現しています。このように、SETDB1は様々な疾患に関連し生体内で重要な役割を担っているにも関わらず、その酵素活性化機構は未だに明らかになっていません。SETDB1は様々な疾患の治療標的であり、酵素活性化機構の解明は、SETDB1の活性を阻害する創薬の応用に繋がります。そこで本研究グループでは、SETDB1の酵素活性化機構を明らかにするための構造解析に向けた、大腸菌内での効率的な活性化型モノユビキチン化SETDB1の調製に取り組みました。
研究内容
立体構造解析に向けて、私たちは大腸菌内でモノユビキチン化SETDB1を調製するために、大腸菌内ユビキチン化プラスミドを構築しました。典型的なタンパク質のユビキチン化には、ユビキチン活性酵素 (E1)、ユビキチン結合酵素 (E2)、ユビキチン連結酵素 (E3) の3つの酵素で段階的に行われます。しかし、SETDB1のユビキチン化にはE3酵素を必要としません。そこで本研究では、E1, UBE2E1 (E2), ユビキチンの3つの遺伝子をコードしたプラスミドを作製しました。このユビキチン化プラスミドと、SETDB1をコードする発現プラスミドを大腸菌内で共発現させた結果、部位特異的にSETDB1のK867のモノユビキチン化に成功しました。また、未修飾のSETDB1とモノユビキチン化SETDB1をそれぞれ精製し、MALDI-TOF/MSを用いてin vitroでSETDB1ヒストンH3メチル化活性を測定しました。未修飾のSETDB1ではヒストンメチル化活性は見られませんでしたが、モノユビキチン化SETDB1ではヒストンメチル化活性が見られました。このことから、大腸菌内ユビキチン化システムにより、酵素活性化型のモノユビキチン化SETDB1の調製に成功し、その有用性を示すことができました。
今後の展望
SETDB1は様々な病気との関連が報告されていますが、未だにその立体構造は未解明です。本研究では大腸菌内ユビキチン化システムでユビキチン化SETDB1の調製に成功したので、構造解析に向けた活性型SETDB1の高純度な精製が可能になると考えられます。今後この技術を活用してユビキチン化SETDB1の構造解析を行い、活性化機構の解明や立体構造に基づいた機能阻害剤の開発が進むことが期待されます。
*本研究は科学研究費補助金 (JP18H02392, 19H05294 and JP19H05741), 公益財団法人武田科学振興財団 (1871140003), 横浜市立大学戦略的研究推進事業 (No. SK201904) の補助を受けて行われました。
*本研究は科学研究費補助金 (JP18H02392, 19H05294 and JP19H05741), 公益財団法人武田科学振興財団 (1871140003), 横浜市立大学戦略的研究推進事業 (No. SK201904) の補助を受けて行われました。
発表雑誌
論文タイトル:Preparation of the ubiquitination-triggered active form of SETDB1 in Escherichia coli for biochemical and structural analyses.
著者:Funyu T, Kanemaru Y, Onoda H, Arita K.
雑誌名:The Journal of Biochemistry, 2021年7月29日オンライン
DOI番号:doi:10.1093/jb/mvab087
著者:Funyu T, Kanemaru Y, Onoda H, Arita K.
雑誌名:The Journal of Biochemistry, 2021年7月29日オンライン
DOI番号:doi:10.1093/jb/mvab087
問い合わせ先
TEL: 045-508-7225, FAX: 045-508-7365
E-mail: aritak@yokohama-cu.ac.jp