構造エピゲノム科学研究室 明石グループの研究成果を掲載
2019.11.12
- TOPICS
- 研究
- 学生の活躍
DNAメチル化サイトの簡便な決定法の開発 (構造エピゲノム科学研究室 明石グループ)
発表者:牛島 弘嗣1 (前期課程1年), 前川 怜奈1 (2018年度前期課程修了), 五十嵐 愛莉1 (2017年度前期課程修了), 明石 知子
⇒Research map
研究分野:構造生物化学
キーワード: DNAメチル化、エピジェネティクス、質量分析、構造解析
1 構造エピゲノム科学研究室 明石グループ
⇒Research map
研究分野:構造生物化学
キーワード: DNAメチル化、エピジェネティクス、質量分析、構造解析
1 構造エピゲノム科学研究室 明石グループ
研究成果のポイント
- 500塩基対程度までの長さの二重鎖DNAのメチル化の状態を迅速かつ正確に決定するための質量分析による手法を開発
- 構造生物学において利用されるDNAの修飾の解析に有効
発表雑誌
論文タイトル:Rapid and definitive analysis of in vitro DNA methylation by nanoelectrospray ionization mass spectrometry
著者:Ushijima H., Maekawa R., Igarashi E., *Akashi S.
雑誌名:Journal of the American Society for Mass Spectrometry (2019) 30, 2335-2346.
著者:Ushijima H., Maekawa R., Igarashi E., *Akashi S.
雑誌名:Journal of the American Society for Mass Spectrometry (2019) 30, 2335-2346.
説明
遺伝情報を担うDNAは、ヒストンタンパク質とヌクレオソームコア粒子を形成して、高次に折りたたまれることで核内に収納されている。
DNAやヒストンタンパク質には様々な修飾が起こることが知られており、この修飾により転写制御が行われている。
DNAでは主にシトシンとグアニンが連なった配列のシトシンへのメチル化が起こる。
DNAのプロモーター領域のCpGアイランドにメチル化が起こると転写が抑制される。
メチル化されたヌクレオソームコア粒子の構造生物学の研究を進めるには、試験管内で修飾されたDNAのメチル化状態を正確に把握する必要がある。
メチル化状態を解析するには、これまでは、メチル化されているとDNA鎖を切断できなくなるメチル化依存性の特定の制限酵素と電気泳動を組み合わせた方法が一般的なものであった。
しかし、この方法では、メチル化されうるすべてのCpG配列を特定の制限酵素が認識する配列に揃える必要があり、解析に用いられるDNA配列に制限がある。
そこで、DNAの配列に非依存的に解析可能な、メチル化前後の質量差(+14 Da/メチル化部位)によってDNA上のメチル化の数を正確かつ迅速に決定する手法を、質量分析を用いて開発した。
DNAやヒストンタンパク質には様々な修飾が起こることが知られており、この修飾により転写制御が行われている。
DNAでは主にシトシンとグアニンが連なった配列のシトシンへのメチル化が起こる。
DNAのプロモーター領域のCpGアイランドにメチル化が起こると転写が抑制される。
メチル化されたヌクレオソームコア粒子の構造生物学の研究を進めるには、試験管内で修飾されたDNAのメチル化状態を正確に把握する必要がある。
メチル化状態を解析するには、これまでは、メチル化されているとDNA鎖を切断できなくなるメチル化依存性の特定の制限酵素と電気泳動を組み合わせた方法が一般的なものであった。
しかし、この方法では、メチル化されうるすべてのCpG配列を特定の制限酵素が認識する配列に揃える必要があり、解析に用いられるDNA配列に制限がある。
そこで、DNAの配列に非依存的に解析可能な、メチル化前後の質量差(+14 Da/メチル化部位)によってDNA上のメチル化の数を正確かつ迅速に決定する手法を、質量分析を用いて開発した。
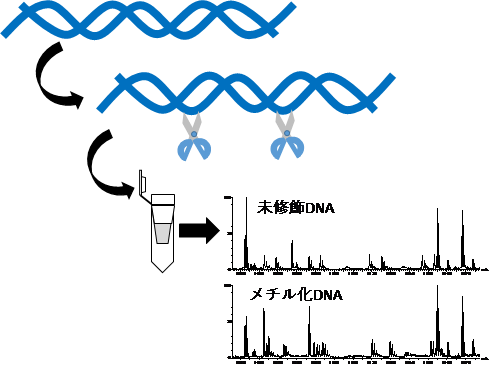 図1. 今回開発した手法の概略 (revised from the original TOC figure in JASMS (2019) 30, 2335-2346.)
図1. 今回開発した手法の概略 (revised from the original TOC figure in JASMS (2019) 30, 2335-2346.)
今回開発した手法の概略を図1に示す。
DNAは多数のリン酸基を持っており、生理的条件下では解離して負の電荷を帯びている。
そのため、正の電荷をもつナトリウムイオンなどの無機塩が付加しやすい。
質量分析では、DNAに無機塩が付加したままで測定すると、付加した無機塩に起因するピークがメチル化によって生じる14Daの質量増加を示すピークの判別を難しくすること、そして様々な長さのDNAを液体クロマトグラフィー(LC)で脱塩・分離するには、条件検討に多くの時間を要することという問題点があった。
そこでメチル化非依存性の制限酵素で断片化した後、LCを用いなくてもエタノール沈殿とゲルろ過スピンカラムによる脱塩を組み合わせて、ほとんどの無機塩を除去しメチル化されたDNAの質量分析を簡便に行えるようになり、メチル化による質量変化を迅速かつ確実に追跡することに成功した。
DNAは多数のリン酸基を持っており、生理的条件下では解離して負の電荷を帯びている。
そのため、正の電荷をもつナトリウムイオンなどの無機塩が付加しやすい。
質量分析では、DNAに無機塩が付加したままで測定すると、付加した無機塩に起因するピークがメチル化によって生じる14Daの質量増加を示すピークの判別を難しくすること、そして様々な長さのDNAを液体クロマトグラフィー(LC)で脱塩・分離するには、条件検討に多くの時間を要することという問題点があった。
そこでメチル化非依存性の制限酵素で断片化した後、LCを用いなくてもエタノール沈殿とゲルろ過スピンカラムによる脱塩を組み合わせて、ほとんどの無機塩を除去しメチル化されたDNAの質量分析を簡便に行えるようになり、メチル化による質量変化を迅速かつ確実に追跡することに成功した。
 図2 メチル化前後の147 bp DNAのマススペクトル(上段:メチル化前、下段:メチル化後) (revised from the original figure in JASMS (2019) 30, 2335-2346.)
図2 メチル化前後の147 bp DNAのマススペクトル(上段:メチル化前、下段:メチル化後) (revised from the original figure in JASMS (2019) 30, 2335-2346.)
図2はメチル化前後の147 bp DNAのマススペクトルである。このようにメチル化が不完全であってもLCでの分離を行わず、迅速に14 Daの質量差を見分けられることがわかる。
問い合わせ先
TEL: 045-508-7217, FAX: 045-508-7362
E-mail: akashi@yokohama-cu.ac.jp